Segmentácia v QuPath
Na vykonanie segmentácie vyšších morfologických štruktúr importujte snímky WSI(Whole Slide Imaging) do projektu QuPath, ktorý ste si stiahli počas inštalácie.
Vytvorte anotácie pre tkanivo a bunky spolu s ich klasifikáciami. Pre tento krok môžete použiť stiahnutý skript.
Na použitie stiahnutého skriptu
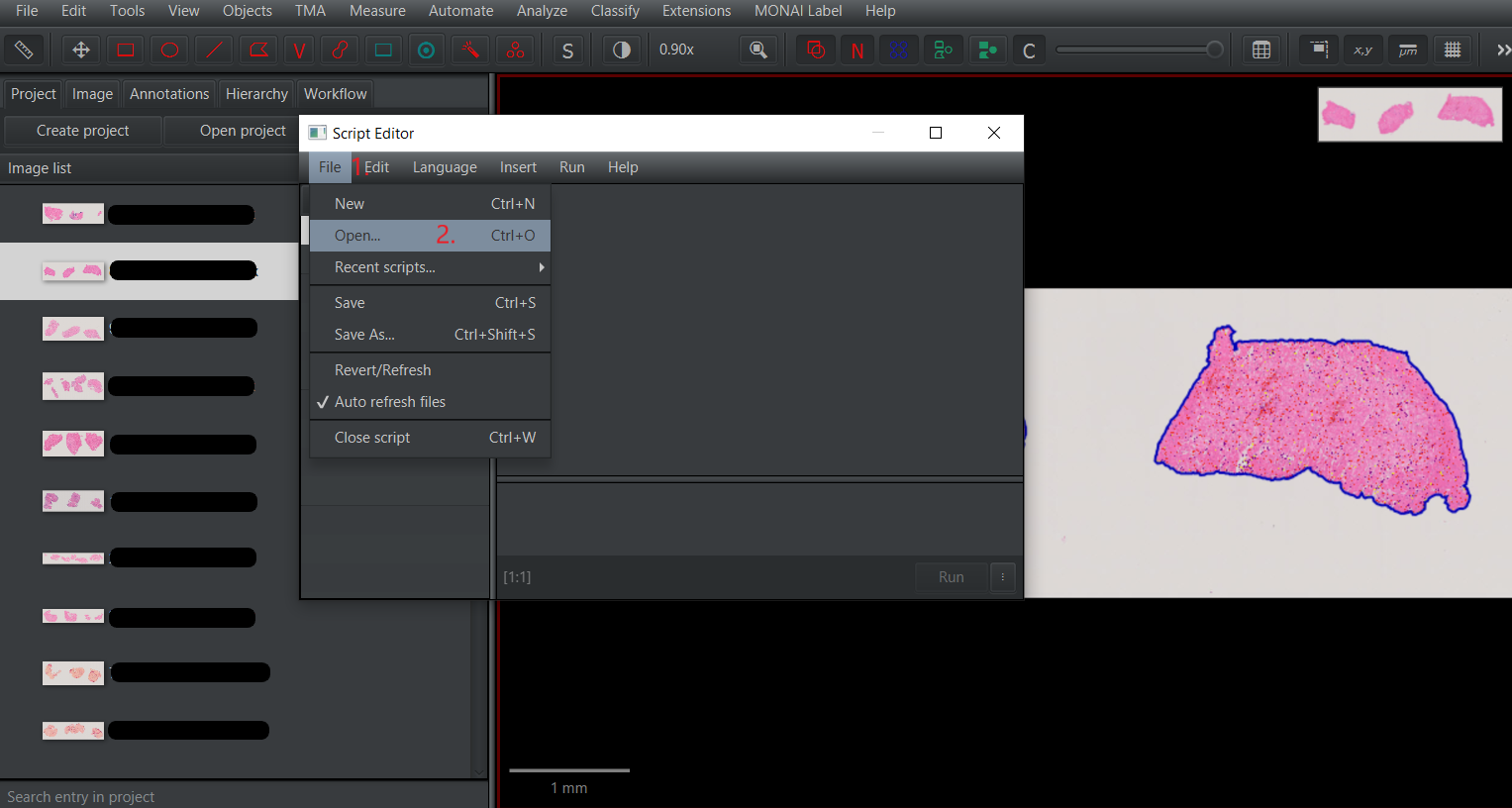
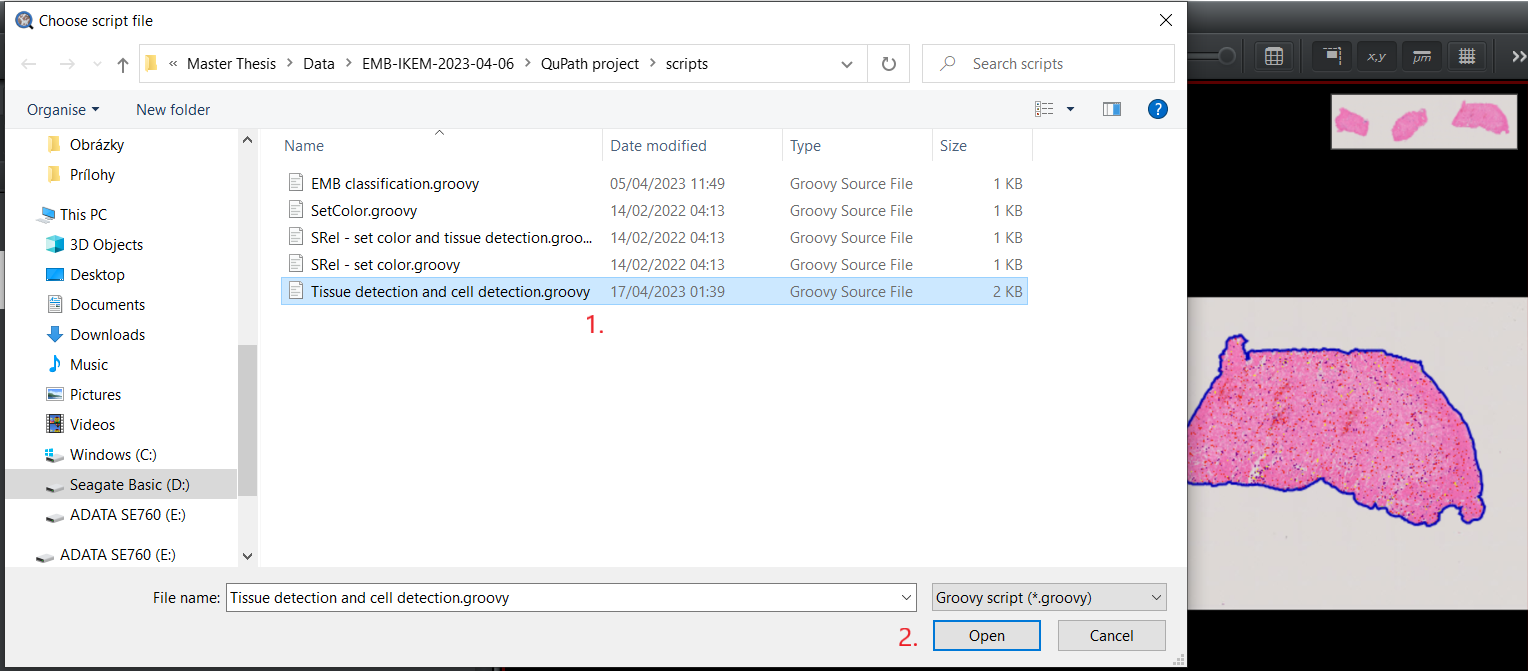
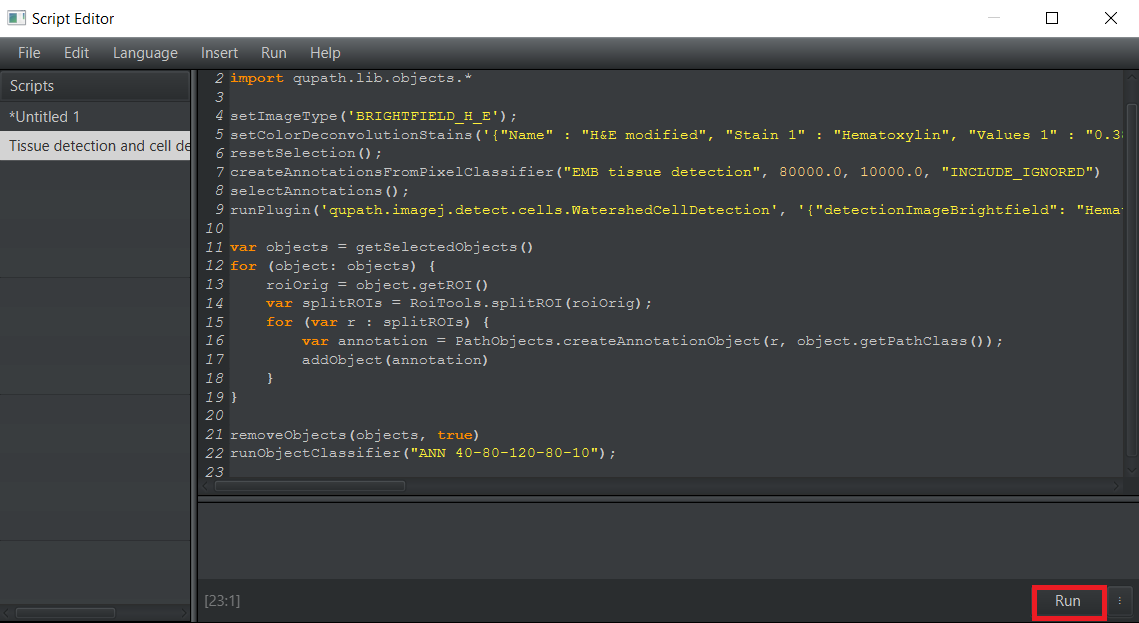
Tissue detection and cell detection.groovyho otvorte v paneliAutomate>Show script editor. Po otvoreníScript editorvyberteFile>Open...a vyberte adresár, kde sa nachádzajú stiahnuté skripty. Vyberte skriptTissue detection and cell detection.groovy. Po načítaní skriptu vyberte možnosťRun, aby ste vykonali segmentáciu tkaniva a buniek a klasifikáciu buniek.1.1 Otvorte
Script editor.
1.2 vyberte adresár so scriptom.
1.3 načítajte vybraný script.
1.4 Spustenie scriptu nad snímkom.
Vyberte oblasť snímku, pre ktorú chcete vykonať predikciu.
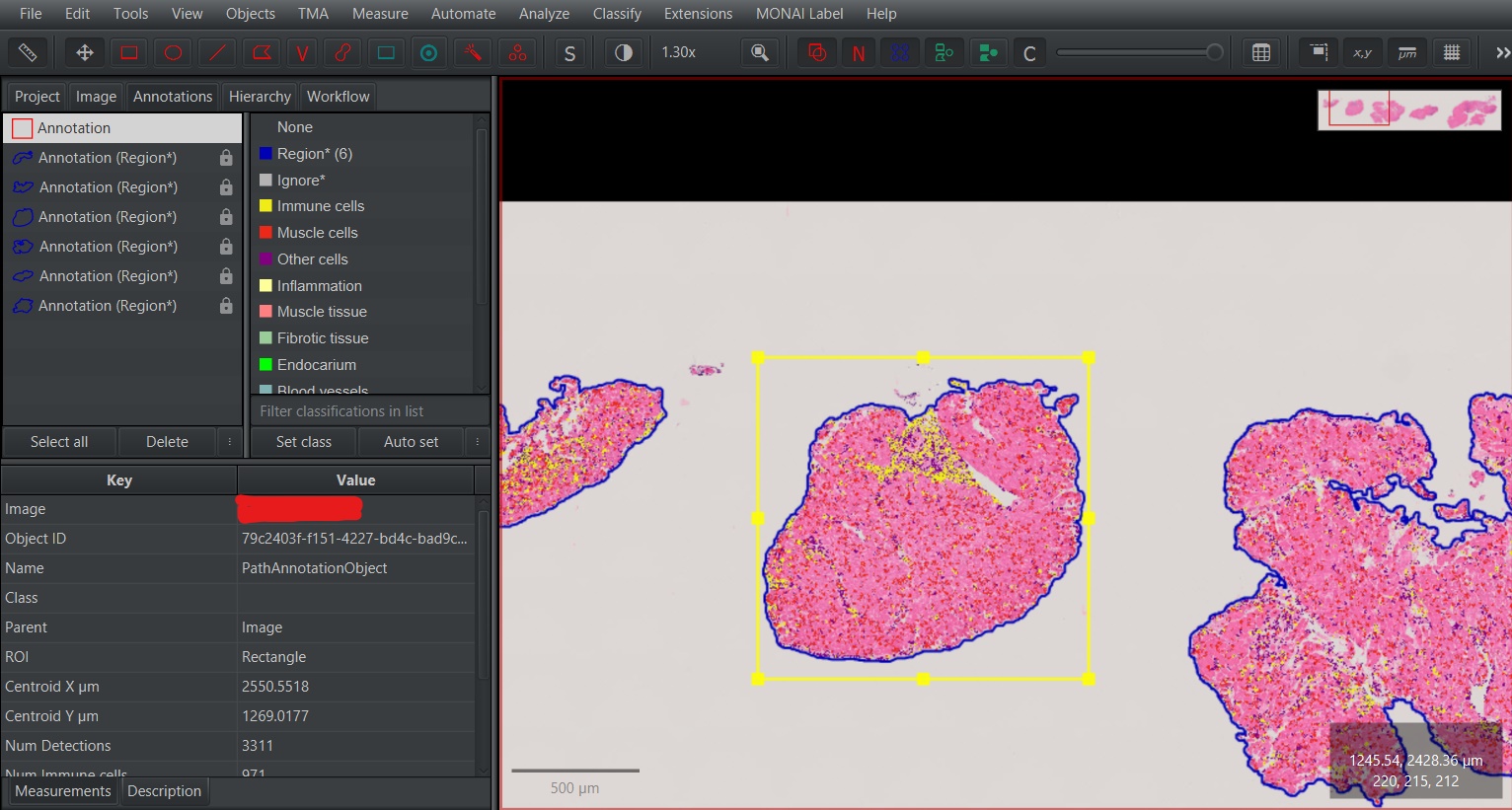
Vyberte
MONAI Label>Create annotations...a potom vyberte model, ktorý chcete použiť pre predikciu v sekciiModel Name.Momentálne dostupné modely sú:
deeplab_structure- DeepLabV3+ na predikciu ciev, zápalu a endokardu v obrázkoch H&Enestedunet_structure- U-Net++ na predikciu ciev, zápalu a endokardu v obrázkoch H&Esrel_segmentation- U-Net na predikciu endokardia v obrázkoch SRel
Po vybratí modelu stlačte
OK.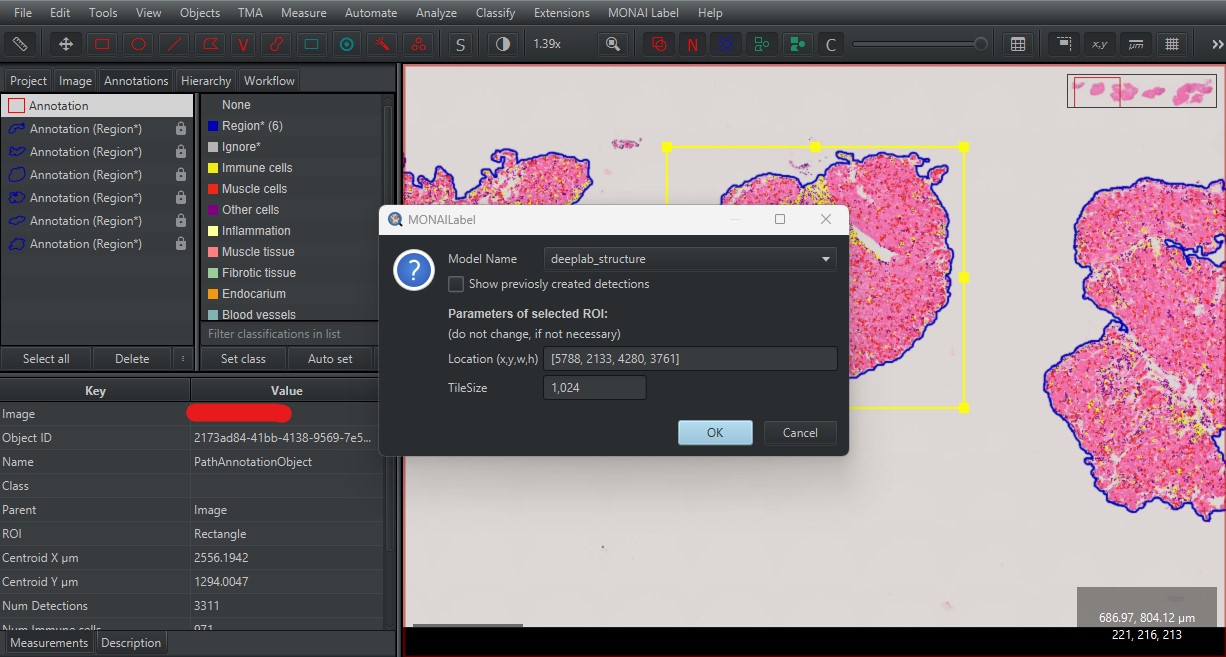
Po dokončení predikcie sú výsledky k dispozícii v QuPath, kde môžete upravovať, mazať alebo manuálne pridávať chýbajúce anotácie.
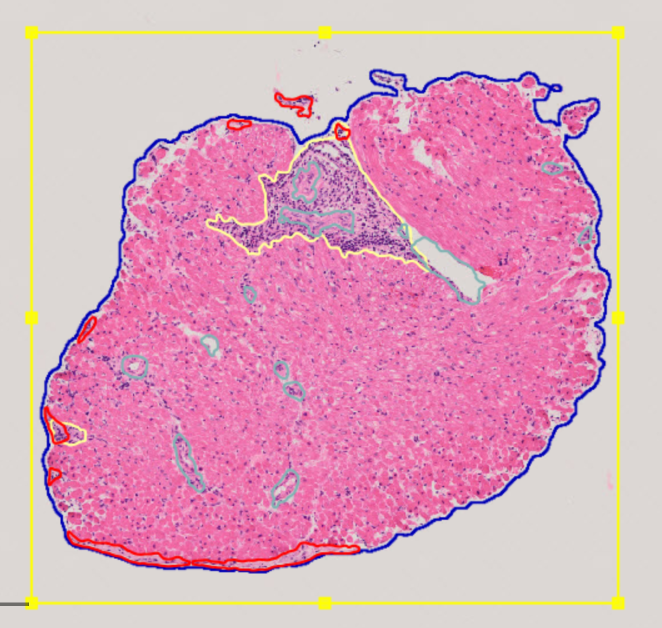
Pre výber anotácií, ktoré chceme vymazať je potrebné spustiť Selection mode výberom možnosti S v paneli. Po povolení Selection mode je možné využiť rôzne spôsoby výberu, či už s využitím obdĺžnika alebo pomocou štetca (brush). Pre zrušenie Selection mode kliknite opätovne možnosť S.
